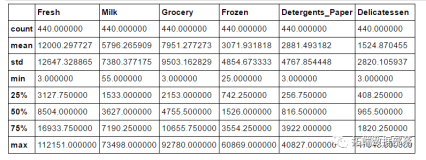
聚类:
在样本中寻找自然集群,事先是不知道存在哪些集群的。聚类是无监督学习,本质是探索数据的结构关系,常用于对客户细分,对文章聚类等
分类:对已经有标签的样本进行分类,已知存在有哪些类别
K-means
原理:事先划定k个点,计算其余点到这k个点的距离,根据距离最短原则划分类别,再重新计算k个类的中心,再进行迭代,直到中心的变化小于设定的阈值
确定聚类数k:K-means算法是无监督学习算法,事先并不知道数据可以聚成几类。使用画图的方式,在高维数据面前也是不可行的。
可以通过设定不同的k值,对应进行k-means聚类。计算k个聚簇内样本点到各自聚簇中心的距离和,把k个聚簇的距离和加总得到总距离。一般而言这个距离会随着k增大而减小,衰减的拐点对应的k值一般而言会是一个比较好的k值。
总距离可以表述为以下公式:
SSE=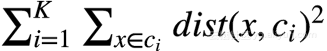
Python实现:
from sklearn.cluster import KMeans
km=KMeans(k)#k为聚簇的数目
km.fit(X)iris上实现K-means
载入数据集
#导入iris的数据集
import pandas
iris =pandas.read_csv('http://archive.ics.uci.edu/ml/machine-learning-databases/iris/iris.data',header=None)
iris.columns=['SepalLengthCm','SepalWidthCm','PetalLengthCm','PetalWidthCm','Species']进行探索性数据分析,根据PetalWidthCm,PetalLengthCm绘制出三个类别的鸢尾花
import seaborn as sns
import matplotlib.pyplot as plt
%matplotlib inline
g=sns.FacetGrid(iris,hue='Species')
g.set(xlim=(0,2.5),ylim=(0,7))
g.map(plt.scatter,'PetalWidthCm','PetalLengthCm').add_legend()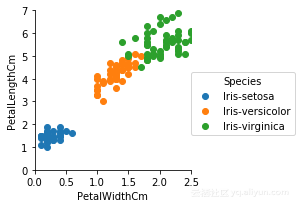
Kmeans聚类
1.设置分类数参数为2
from sklearn.cluster import KMeans
#选取iris聚类特征
X=iris[['PetalWidthCm','PetalLengthCm']]
#设定模型参数
km=KMeans(2)
#训练模型
km.fit(X)
#得到聚类结果
iris['cluster_k2']=km.predict(X)
km.predict(X)
array([1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0])
g=sns.FacetGrid(iris,hue='cluster_k2')
g.set(xlim=(0,2.5),ylim=(0,7))
g.map(plt.scatter,'PetalWidthCm','PetalLengthCm').add_legend()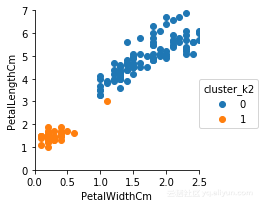
设置分类数参数为3
from sklearn.cluster import KMeans
#选取iris聚类特征
X=iris[['PetalWidthCm','PetalLengthCm']]
#设定模型参数
km=KMeans(3)
#训练模型
km.fit(X)
#得到聚类结果
iris['cluster_k2']=km.predict(X)
km.predict(X)
g=sns.FacetGrid(iris,hue='cluster_k2')
g.set(xlim=(0,2.5),ylim=(0,7))
g.map(plt.scatter,'PetalWidthCm','PetalLengthCm').add_legend()
#看看和之前的绘图结果有什么样的区别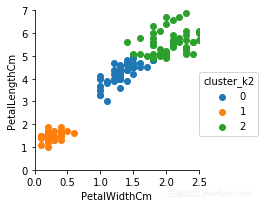
很ok,看看和一开始的那张图(也就是正确分类的图)比较一下,是不是很相似?
K-means算法的局限
K-means算法适用于数据集呈现出类圆形、球形分布的,如果数据没有呈现出这种规律,很可能聚类的效果会是很差的
DBSCAN
DBSCAN,全称是Density-Based Spatial Clustering of Applications with Noise,是一种基于密度的聚类方法
原理:根据$\epsilon$和min_samples把数据点分为三类点,一类是CORE(图中红色点):周围$\epsilon$距离内有大于或等于min_sample个样本点; REACHABLE(图中蓝色点):周围$\epsilon$距离内的样本点数量小于min_sample,但是可以被CORE点覆盖的点(也就是在CORE点以$\epsilon$为半径范围内的点) ; OUTLIER(图中蓝色):异常点,不属于任何一个类别
预先需要给定的参数是:$\epsilon$、min_samples,且对参数的选择非常敏感
- CORE点需要满足的条件是范围内的数据点大于或等于min_samples
- REACHABLE点是被纳入CORE点范围的点,但本身不满足作为CORE点的条件
- OUTLIER:除开以上两类以外的所有数据点
如图:
Python实现:
from sklearn.cluster import DBSCAN
dbscan=DBSCAN(eps=0.3,min_samples=10)
dbscan.fit(X)
dbscan.labels_array([ 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0,
0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1,
1, 1, 1, 1, 1, 1, -1, 1, 1, -1, 1, 1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, -1, 1, 1, 1, 1, 1,
1, 1, 1, 1, 1, 1, 1, 1, -1, 1, 1, 1, 1, -1, 1, -1, 1,
1, -1, 1, -1, 1, -1, -1, -1, 1, 1, 1, 1, -1, 1, 1, -1, -1,
1, 1, 1, -1, 1, 1, -1, 1, 1, 1, -1, -1, -1, 1, 1, -1, -1,
1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1], dtype=int64)
iris上实现DBSCAN
生成数据集
from sklearn import datasets
from pandas import DataFrame
noisy_circles=datasets.make_circles(n_samples=1000,factor=.5,noise=.05)
print(noisy_circles)
df=DataFrame()
df['x1']=noisy_circles[0][:,0]
df['x2']=noisy_circles[0][:,1]
df['label']=noisy_circles[1]
df.sample(10)(array([[ 0.67533655, -0.68506843],
[ 0.45998261, 0.21745649],
[ 0.89143489, -0.06072569],
...,
[ 0.5703842 , 0.87910732],
[ 0.5663019 , -0.75688068],
[ 1.03654422, 0.05237379]]), array([0, 1, 0, 0, 1, 1, 0, 0, 0, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0,
1, 0, 0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 1, 1, 0, 1, 1, 1, 1, 0, 0,
1, 1, 1, 0, 1, 0, 1, 1, 0, 1, 1, 0, 0, 1, 1, 1, 0, 0, 0, 1, 0, 0, 0,
1, 1, 1, 1, 1, 0, 0, 1, 1, 0, 1, 1, 0, 0, 1, 1, 0, 0, 1, 1, 0, 1, 1,
0, 1, 1, 0, 1, 0, 0, 0, 1, 0, 1, 1, 0, 0, 0, 1, 1, 1, 1, 1, 0, 1, 0,
0, 1, 0, 0, 1, 0, 1, 0, 1, 0, 1, 0, 0, 1, 1, 0, 1, 0, 1, 1, 0, 0, 0,
0, 0, 1, 1, 0, 0, 0, 0, 1, 1, 1, 0, 1, 1, 1, 0, 0, 0, 1, 0, 0, 1, 1,
0, 0, 0, 1, 1, 0, 1, 1, 1, 1, 1, 1, 1, 1, 0, 0, 0, 1, 1, 1, 1, 0, 0,
1, 0, 1, 1, 1, 0, 0, 0, 0, 0, 1, 1, 0, 1, 0, 1, 1, 1, 1, 1, 1, 0, 1,
0, 1, 1, 0, 1, 0, 0, 0, 0, 0, 0, 1, 0, 0, 0, 1, 1, 1, 1, 0, 1, 1, 1,
1, 1, 1, 1, 1, 0, 1, 1, 0, 0, 0, 1, 1, 0, 0, 1, 1, 0, 1, 0, 1, 0, 1,
1, 0, 1, 0, 1, 0, 1, 0, 0, 0, 1, 0, 0, 1, 1, 1, 1, 0, 0, 1, 0, 0, 0,
1, 1, 0, 0, 1, 1, 0, 0, 0, 1, 1, 0, 0, 1, 0, 1, 0, 1, 1, 0, 1, 0, 0,
1, 1, 1, 1, 0, 0, 1, 0, 0, 0, 1, 1, 1, 0, 1, 0, 0, 0, 1, 0, 0, 0, 0,
0, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0, 1, 0, 1, 1, 1, 1, 0, 0, 0, 1, 0, 1,
0, 0, 0, 1, 0, 0, 1, 1, 0, 1, 0, 1, 1, 0, 0, 1, 0, 1, 1, 0, 0, 1, 1,
1, 1, 0, 0, 0, 1, 1, 0, 0, 1, 0, 0, 1, 1, 0, 0, 0, 1, 0, 0, 0, 1, 1,
1, 1, 1, 1, 1, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 1, 1, 1, 0, 0, 1, 1, 0,
0, 0, 1, 0, 0, 1, 1, 1, 0, 1, 0, 1, 0, 0, 0, 1, 0, 0, 0, 0, 0, 1, 1,
1, 0, 1, 1, 1, 1, 0, 0, 1, 0, 1, 1, 1, 0, 1, 0, 0, 0, 0, 1, 1, 0, 0,
1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 0, 1, 0, 1, 0, 1, 1, 1, 0, 1, 1, 0, 0,
0, 0, 1, 1, 0, 0, 1, 0, 1, 1, 1, 1, 1, 1, 0, 1, 0, 0, 0, 0, 0, 0, 0,
1, 1, 0, 1, 0, 1, 1, 0, 0, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0, 1, 1, 0, 1,
0, 1, 0, 0, 1, 0, 1, 0, 1, 1, 1, 1, 0, 1, 1, 1, 0, 0, 1, 0, 0, 1, 1,
1, 1, 0, 1, 0, 1, 0, 1, 1, 1, 1, 1, 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 1,
1, 1, 0, 1, 1, 1, 0, 0, 1, 1, 1, 0, 0, 1, 1, 0, 1, 0, 1, 1, 0, 0, 1,
1, 0, 0, 1, 1, 1, 1, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 1, 1, 1, 0, 1,
0, 1, 1, 1, 1, 0, 1, 1, 0, 0, 1, 1, 0, 1, 0, 1, 1, 0, 1, 1, 1, 1, 0,
0, 0, 1, 0, 1, 1, 1, 0, 1, 0, 0, 1, 1, 0, 0, 1, 0, 0, 0, 0, 1, 0, 1,
0, 1, 0, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, 1, 1, 0, 0, 0, 0, 0, 1,
0, 0, 0, 0, 1, 1, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0, 1, 0, 1, 0, 0, 0, 0,
0, 0, 0, 1, 1, 0, 1, 1, 1, 0, 0, 0, 0, 1, 0, 1, 0, 1, 0, 0, 1, 0, 1,
0, 0, 1, 0, 0, 1, 1, 0, 1, 1, 0, 0, 0, 1, 1, 1, 0, 1, 0, 0, 1, 0, 0,
1, 1, 0, 0, 1, 0, 0, 1, 1, 0, 1, 1, 1, 0, 0, 0, 1, 0, 0, 1, 1, 1, 0,
1, 0, 1, 1, 1, 1, 1, 0, 1, 0, 1, 0, 1, 1, 0, 1, 1, 0, 1, 1, 0, 0, 0,
1, 0, 0, 0, 0, 0, 0, 0, 1, 1, 0, 0, 0, 1, 0, 0, 1, 0, 1, 1, 1, 1, 0,
0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 1, 0, 0, 1, 1, 1, 0, 0, 1, 1, 1, 1,
1, 0, 1, 1, 0, 0, 1, 0, 0, 1, 1, 0, 0, 0, 0, 1, 1, 1, 0, 0, 1, 0, 1,
1, 1, 1, 1, 0, 1, 0, 1, 0, 0, 1, 0, 1, 0, 1, 1, 0, 0, 1, 1, 0, 1, 0,
0, 0, 0, 1, 1, 1, 0, 1, 0, 1, 0, 0, 0, 1, 0, 0, 1, 0, 0, 0, 0, 0, 0,
1, 0, 1, 1, 1, 0, 1, 1, 1, 1, 0, 1, 1, 0, 0, 0, 1, 0, 1, 1, 1, 1, 0,
0, 0, 0, 1, 1, 1, 1, 1, 0, 1, 1, 1, 0, 0, 0, 0, 1, 0, 1, 0, 1, 1, 1,
1, 0, 0, 0, 1, 1, 0, 1, 1, 1, 0, 1, 1, 0, 1, 0, 1, 1, 1, 1, 1, 0, 1,
0, 1, 0, 1, 0, 0, 0, 1, 0, 0, 0], dtype=int64))
| x1 | x2 | label | |
|---|---|---|---|
| 554 | 0.951496 | -0.573552 | 0 |
| 509 | 0.176190 | 0.435655 | 1 |
| 612 | -0.267315 | -0.927010 | 0 |
| 983 | 0.476075 | 0.092146 | 1 |
| 729 | 0.795222 | -0.474817 | 0 |
| 928 | -0.351494 | -0.449584 | 1 |
| 313 | 0.493773 | 0.280621 | 1 |
| 204 | -0.073940 | -0.493139 | 1 |
| 984 | 0.080212 | -0.497828 | 1 |
| 796 | 0.725219 | 0.750190 | 0 |
进行探索性数据分析
import seaborn as sns
import matplotlib.pyplot as plt
%matplotlib inline
g=sns.FacetGrid(df,hue='label')
g.map(plt.scatter,'x1','x2').add_legend()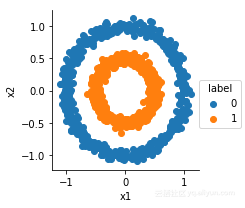
DBSCAN
from sklearn.cluster import DBSCAN
dbscan=DBSCAN(eps=0.2,min_samples=10)
X=df[['x1','x2']]
dbscan.fit(X)
df['dbscan_label']=dbscan.labels_
g=sns.FacetGrid(df,hue='dbscan_label')
g.map(plt.scatter,'x1','x2').add_legend()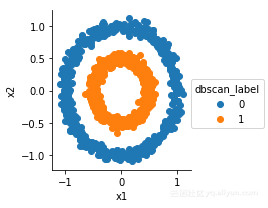
ok!很完美!
但是,DBSCAN对参数设置特别敏感
比如,我们可以尝试修改epsilon
from sklearn.cluster import DBSCAN
dbscan=DBSCAN(eps=0.1,min_samples=10)
X=df[['x1','x2']]
dbscan.fit(X)
df['dbscan_label']=dbscan.labels_
g=sns.FacetGrid(df,hue='dbscan_label')
g.map(plt.scatter,'x1','x2').add_legend()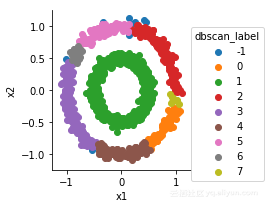
from sklearn.cluster import DBSCAN
dbscan=DBSCAN(eps=0.3,min_samples=10)
X=df[['x1','x2']]
dbscan.fit(X)
df['dbscan_label']=dbscan.labels_
g=sns.FacetGrid(df,hue='dbscan_label')
g.map(plt.scatter,'x1','x2').add_legend()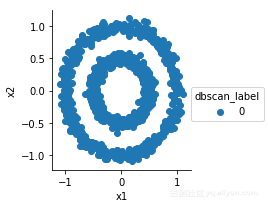
K-means
K-means聚类步骤:
- Step1: 随机选择k个质心(即k个类);
- Step2: 计算每一个样本点到这些质心的距离,把样本点划分给距离最短的质心,从而把所有的样本点划分为k类,形成k个聚簇;
- Step3: 对于每个类,重新计算聚簇的中心,确定该类的质心;
- Step4: 若收敛,则结束;否则转到Step2.
K-means缺点:
- K-means算法的随机性主要在于初始点的选取,且对聚类中心的初始选择比较敏感,初始点的选择会影响最终的聚类效果
- k值需要首先人工确定(启发式)。
- 对噪声比较敏感